Bei jeder Blutabnahme werden sie untersucht, um Informationen über physiologische Prozesse als eine Art »Wasserstandsmeldung« zu erhalten. Biomarker oder Surrogatparameter sind nicht nur wichtige Moleküle zur Diagnosestellung, sondern werden genauso zur Überwachung nach einer Erkrankung oder zur Früherkennung von Krankheiten eingesetzt. Forscher*innen und Mediziner*innen treffen sich hier an einem Schnittpunkt ihrer Arbeit und können gemeinsam biointelligente Konzepte erarbeiten. Zum Start dieser Blog-Serie reisen wir in die molekularen Tiefen des Körpers um ein Gefühl für Biomarker zu erhalten.
Schon vor knapp 2500 Jahren sagte einst Konfuzius »Der Weg ist das Ziel« und in wohl keinem anderen Bereich gilt das so sehr wie in der medizinischen Diagnostik. Es kann nur korrekt therapiert werden sofern man genau weiß, was es zu behandeln gilt. Neben bildgebenden Verfahren zur Diagnostik wie MRT oder CT sind Biomarker ein wichtiger Indikator, sei es zur Krankheitsdiagnostik, zum Monitoring des Heilungsverlaufes oder als routinemäßige Überwachung.
Was sind Biomarker und Surrogatparameter? – oder: Keine Angst vor Abkürzungen
Wer kennt sie nicht, die Liste nach der Blutabnahme für die Laboruntersuchung: GPT, GGT, Kreatinin, Troponin, CRP, HDL, HbA1c – eine schier endlose Liste mit Zahlen und Normwerten. All diese Parameter helfen Mediziner*innen und Forscher*innen dabei, ein Gefühl für ablaufende (patho-) physiologische Prozesse im Körper zu gewinnen. Doch was bedeuten sie und was sind Biomarker sowie Surrogatparameter?
Unter dem Begriff Biomarker versteht man Moleküle, die prognostische oder diagnostische Aussagekraft für einen biologischen oder krankhaften Prozess haben und daher als frühe Hinweise für Krankheiten dienen. Als Beispiel hierfür steht das oben erwähnte GPT. Es ist eine Abkürzung für das Enzym Glutamat-Pyruvat-Transaminase. Dieses Enzym wird bei Schädigungen aus Leberzellen ins Blut abgegeben und deutet dementsprechend auf Lebererkrankungen oder -schäden hin. Das Molekül CRP (C-reaktives Protein), als weiteres Beispiel, wird vom Körper als Reaktion auf eine Infektion erhöht ausgeschüttet, um aktiv die Infektion zu bekämpfen und Erreger abzutöten. Mediziner*innen machen sich dieses Wissen zu Nutzen und können durch die ermittelten Werte Rückschlüsse auf eine aktive Infektion oder Entzündungsreaktion schließen.
Ein Surrogatparameter dient als Ersatzgröße oder Ableitung für etwas, das sonst nur schwerlich oder gar nicht gemessen werden kann. Der Hba1c-Wert – vereinfacht gesagt die Menge an Zucker-gebundenem Hämoglobin − wird zum Beispiel bei Diabetes-Patient*innen gemessen, um Rückschlüsse auf die korrekte Dosis/Einstellung an Insulin innerhalb der letzten zwei bis drei Monate zu geben und ist dabei aussagekräftiger als tägliches »Zucker messen«. Bei einem Surrogatparameter besteht ein statistischer, jedoch nicht zwingend ein kausaler Zusammenhang. Als Beispiel hierfür dient die Steigerung der Herzfrequenz bei Angst. Es zeigt sich schon, dass nicht jedes Molekül die Fähigkeit hat, als geeigneter Biomarker/Surrogatparameter klinisch verwendet zu werden.
Anforderungen an Biomarker und deren Wichtigkeit – oder: »Entscheidend is‘ auf’m Platz«
Als bekennendem Fußballfan ist mir dieser Satz von Alfred Preißler nur zu gut bekannt – und ebenfalls auch auf Biomarker umzumünzen. Was in der Theorie gut ist, muss noch lange nicht in der Praxis am besten geeignet sein. Denn jeder Biomarker − egal ob diagnostischer, prognostischer oder prädiktiver Art – hat bestimmte Anforderungen zu erfüllen, um klinisch relevant zu sein. Einerseits muss ein Biomarker natürlich spezifisch sein und klar assoziiert mit der Fragestellung. Er sollte so zum Beispiel zu einem hohen Prozentsatz (>90 %) die Erkrankung erkennen können. Darüber hinaus ist es wichtig, dass er eine geringe falsch-positive Rate aufweist, das heißt sich kaum irrt und fälschlicherweise eine Erkrankung vermuten lassen. Diese sogenannte Sensitivität und Spezifität werden in klinischen Studien validiert.
Jedoch spielen auch noch andere Aspekte eine Rolle. Behandelnde Ärzt*innen sind angehalten, bei der Wahl ihrer Methoden schonend mit Patient*innen umzugehen, um keine unnötigen oder gefährlichen Eingriffe durchzuführen. Deswegen sollte ein Biomarkermolekül möglichst schonend gewonnen werden (minimal/non-invasiv). So kann ein molekularer Biomarker für eine Hirnerkrankung zum Beispiel perfekt zur Vorhersage einer Erkrankung geeignet sein – wenn er nicht durch bildgebende oder minimal/nicht-invasive Verfahren gemessen werden kann, ist er für eine Früherkennung oder Monitoring nicht geeignet. Oder wem könnte man bei einer routinemäßigen Vorsorgeuntersuchung erzählen, dass jetzt der Schädel aufgeschnitten werden muss für eine Gehirnprobe? Somit ist für den Erfolg eines Biomarkers ein einfaches Entnahmeprotokoll sowie dessen Stabilität und Weiterverwendbarkeit ebenso entscheidend. Wenn dieses Verfahren dazu auch noch günstig ist, stehen die Chancen für eine Verwendung in klinischen Fragestellung gut.
Wir von der In-vitro-Diagnostik am Fraunhofer IGB machen uns den Biomarker zellfreie DNA (cell-free DNA – cfDNA) zu Nutze. Mittels einer sogenannten Liquid Biopsy (siehe Abbildung 1) entnehmen wir Patient*innen eine geringe Menge Blut und isolieren daraus die cfDNA. Diese Methode ist nicht nur günstig und minimal-invasiv: Durch die einfache Lagerung des aus dem Blut gewonnenen Plasmas bei -80 °C und durch unkomplizierte, schnelle Protokolle werden bereits viele Anforderungen erfüllt und die Methode ist auch molekularbiologisch mittels biointelligenter Verfahren sehr gut geeignet für unsere Fragestellungen. Doch was ist cfDNA eigentlich?
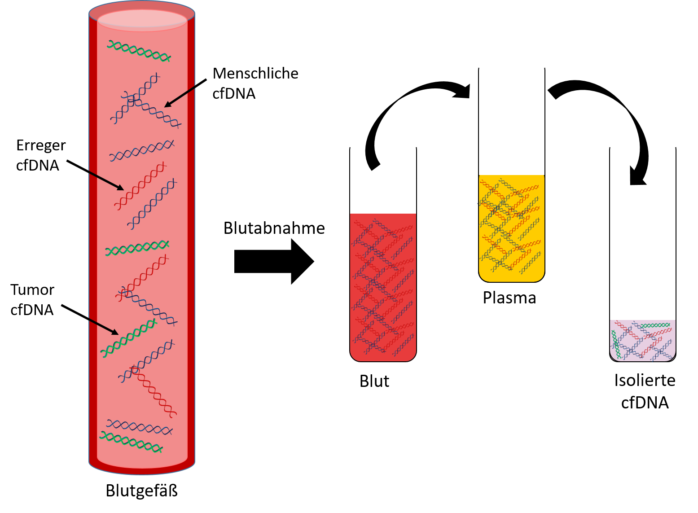
Zellfreie DNA als Biomarker in Forschung und Diagnostik – oder: Gib mir dein Blut und ich zeig dir, was du hast
Wie es der Name bereits andeutet, befindet sich cfDNA außerhalb von Zellen und ist dadurch unter anderem im Blut oder Urin nachzuweisen. Entdeckt vor über 70 Jahren von den Forschern Mandel & Métais, ist cfDNA gerade in den letzten Jahren in den Mittelpunkt der molekularen Diagnostik und Krankheitsprognose gerückt. Alle kernhaltigen Zellen im Körper setzen DNA frei – neben gesunden und zerstörten körpereigenen Zellen auch Krebszellen oder Mikroorganismen wie Bakterien. Dabei ist cfDNA nicht nur eine Art Überbleibsel eines zellulären Abbaumechanismus, sondern wird auch aktiv abgegeben, mutmaßlich als Botschafter zwischen verschiedenen Körperzellen. Es ist darüber hinaus bekannt, dass cfDNA von bestimmten Immunzellen, den sogenannten neutrophilen Granulozyten, in der Abwehr von Krankheitserregern aktiv freigesetzt wird. Zusammen mit anderen ausgeschütteten Proteinen bilden sich sogenannte NETs (neutrophil extracellular traps) – molekulare Fangnetze im Blut, die Krankheitserreger binden und unschädlich machen. Sowohl bei Infektionen als auch nach sportlicher Betätigung, einem Trauma oder im Alter ist die Konzentration erhöht.
Wir von der In-vitro-Diagnostik am Fraunhofer IGB sind im Speziellen an der Erkennung von Pankreaskrebs und der Erregerdiagnostik bei Sepsis interessiert. Wie bereits oben beschrieben, isolieren wir mittels Liquid Biopsy (Abbildung 1) vollautomatisiert die cfDNA aus dem Blut. Da neben körpereigener, gesunder cfDNA auch Sepsiserreger oder Krebszellen ihre DNA in das Blut abgeben, isolieren wir auch diese dadurch. Aufgrund einzigartiger molekularer DNA-Sequenzen eines jeden Erregers oder Krebstyps können wir diese genetischen Spuren mithilfe biointelligenter Methoden erkennen und durch bioinformatische Analyse bestimmen, um zum Beispiel eine Diagnose für einen verursachenden Sepsiserreger zu stellen. Wie genau man den Erregern oder Tumoren mittels dem Biomarker cfDNA auf die Spur kommt, ist eine Geschichte für den zweiten Teil dieser Blog Serie – bis zum nächsten Mal, wenn es wieder heißt: Auf den Spuren von Sherlock Holmes: Wenn Forschende zu Detektiven werden.
Interesse geweckt?
Das Innovationsfeld In-vitro-Diagnostik steht für die Etablierung, Entwicklung und Verwertung diagnostischer Verfahren für biologische und medizinische Anwendungsgebiete. Dabei werden innovative Verfahren der Molekularbiologie, Biochemie und der Bioinformatik entwickelt, kombiniert und angewendet. Durch unsere jahrelangen Erfahrungen im Bereich Erregerdiagnostik bei Sepsis besitzen wir eine weitreichende Expertise auf diesem Themengebiet und wollen diese stetig in anderen Bereichen, wie zum Beispiel der Pankreaskrebsdiagnostik, weiterentwickeln. Die Next-Generation-Sequencing-Technologie (NGS) zur Hochdurchsatzsequenzierung von Nukleinsäuren stellt dabei eine der zentralen Technologieplattformen innerhalb des Innovationsfelds dar. So nutzen wir NGS auch für die De-novo-Sequenzierung von Bakterien und Pilzen, Transkriptionsprofilanalysen, die Identifizierung relevanter Gene oder in der Corona-Diagnostik ein. Wenn Sie mehr darüber erfahren möchten, dann treten Sie gerne mit uns im Kontakt. #WeKnowHow

